Try in Colab
- Weights & Biasesのrunを初期化し、再現性を確保するためにrunに関連するすべての設定を同期。
- MONAIトランスフォームAPI:
- 辞書形式のデータ用のMONAIトランスフォーム。
- MONAIの
transformsAPIに従った新しいトランスフォームの定義方法。 - データ拡張のための強度をランダムに調整する方法。
- データのロードと可視化:
- メタデータで
Nifti画像をロードし、画像のリストをロードしてスタック。 - IOとトランスフォームをキャッシュしてトレーニングと検証を加速。
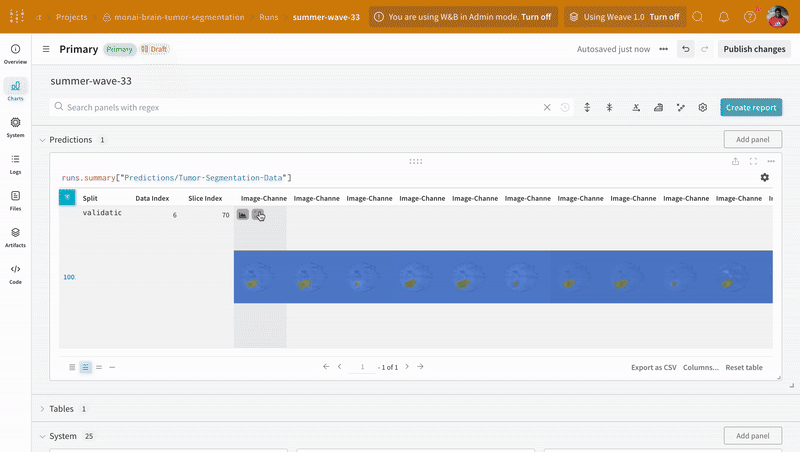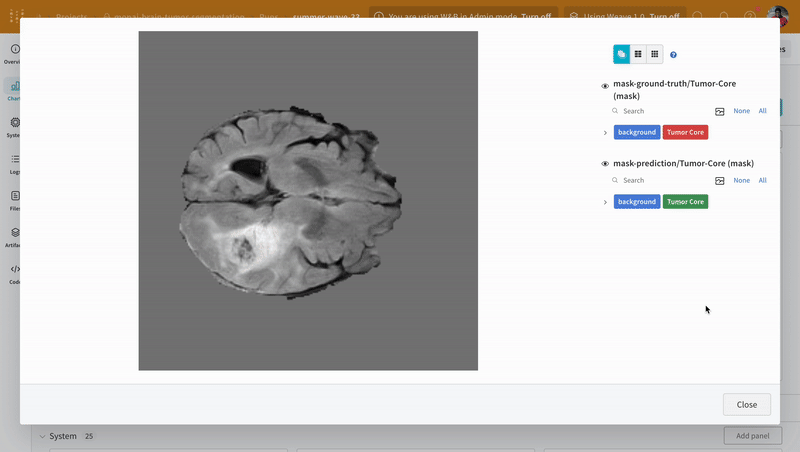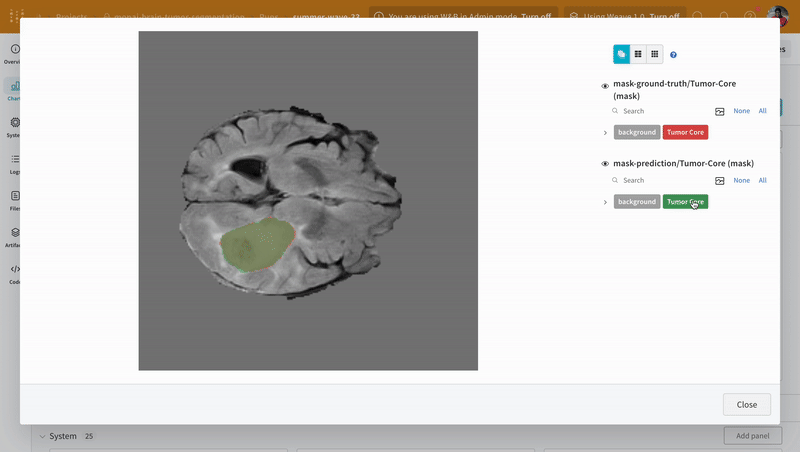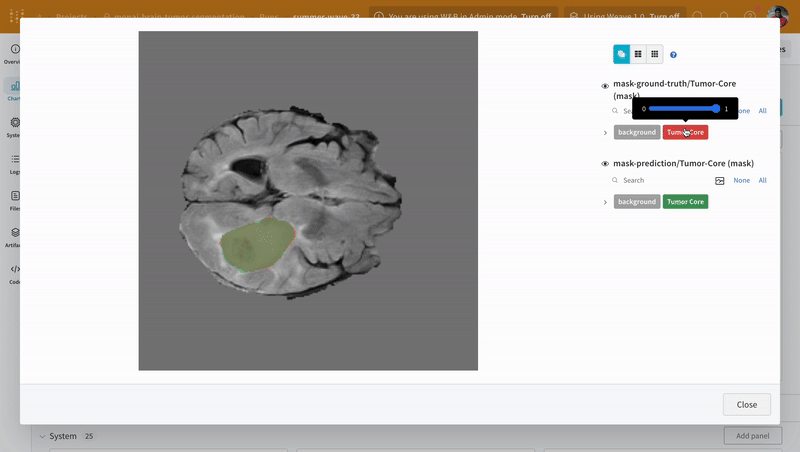
wandb.TableとWeights & Biasesでインタラクティブなセグメンテーションオーバーレイを用いてデータを可視化。
- メタデータで
- 3D
SegResNetモデルのトレーニング- MONAIの
networks,losses,metricsAPIsを使用。 - PyTorchトレーニングループを使用して3D
SegResNetモデルをトレーニング。 - Weights & Biasesを使用してトレーニングの実験管理を追跡。
- Weights & Biases上でモデルのチェックポイントをモデルアーティファクトとしてログとバージョン管理。
- MONAIの
wandb.TableとWeights & Biasesでインタラクティブなセグメンテーションオーバーレイを使用して、検証データセット上の予測を可視化して比較。
セットアップとインストール
まず、MONAIとWeights & Biasesの最新バージョンをインストールします。W&B Runの初期化
新しいW&B runを開始して実験を追跡します。データのロードと変換
ここでは、monai.transforms APIを使用して、マルチクラスラベルをワンホット形式でのマルチラベルセグメンテーションタスクに変換するカスタムトランスフォームを作成します。
データセット
この実験で使用されるデータセットは、http://medicaldecathlon.com/ から入手可能です。マルチモーダルおよびマルチサイトMRIデータ(FLAIR, T1w, T1gd, T2w)を使用して、膠芽腫、壊死/活動中の腫瘍、および浮腫をセグメント化します。データセットは750個の4Dボリューム(484 トレーニング + 266 テスト)で構成されています。DecathlonDataset を使用してデータセットを自動的にダウンロードし、抽出します。MONAI CacheDataset を継承し、cache_num=N を設定してトレーニングのために N アイテムをキャッシュし、メモリサイズに応じてデフォルト引数を使用して検証のためにすべてのアイテムをキャッシュできます。
注意:
train_transform を train_dataset に適用する代わりに、val_transform をトレーニングおよび検証データセットの両方に適用します。これは、トレーニングの前に、データセットの2つのスプリットからのサンプルを視覚化するためです。データセットの可視化
Weights & Biasesは画像、ビデオ、オーディオなどをサポートしています。リッチメディアをログに取り込み、結果を探索し、run、モデル、データセットを視覚的に比較できます。セグメンテーションマスクオーバーレイシステムを使用してデータボリュームを可視化します。 テーブルにセグメンテーションマスクをログに追加するには、テーブル内の各行にwandb.Image オブジェクトを提供する必要があります。
次の擬似コードは、その例です。
wandb.Table オブジェクトと関連するメタデータを受け取り、Weights & Biases ダッシュボードにログされるテーブルの行を埋めるユーティリティ関数を作成します。
wandb.Table オブジェクトと、それが含む列を定義し、データ可視化を使用してそれを埋めます。
train_dataset とval_dataset をループして、データサンプルの可視化を生成し、ダッシュボードにログを取るためのテーブルの行を埋めます。
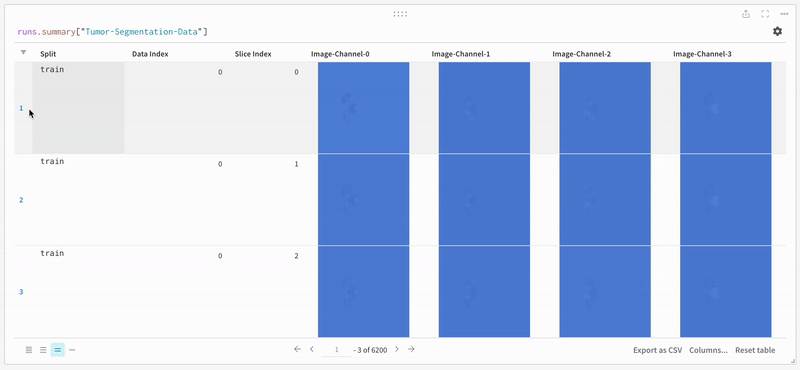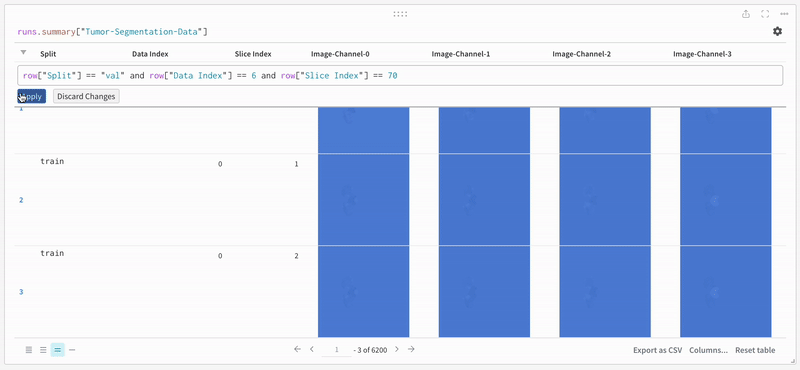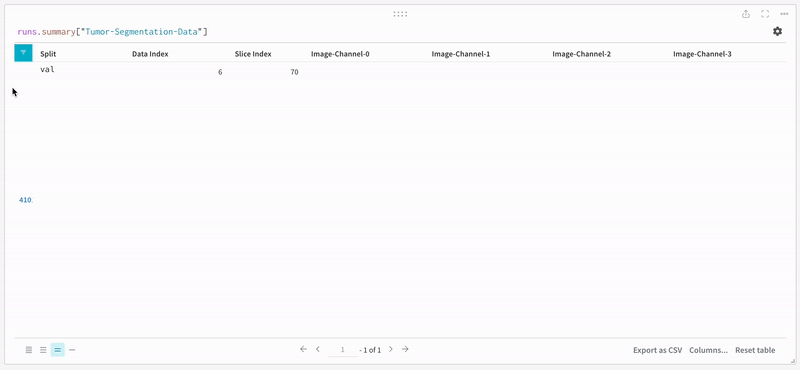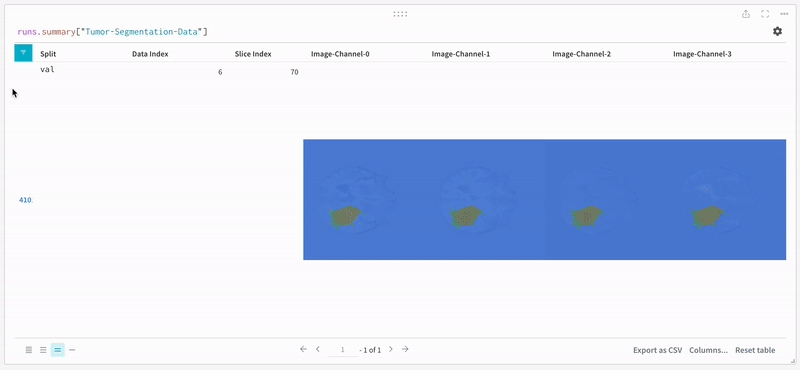
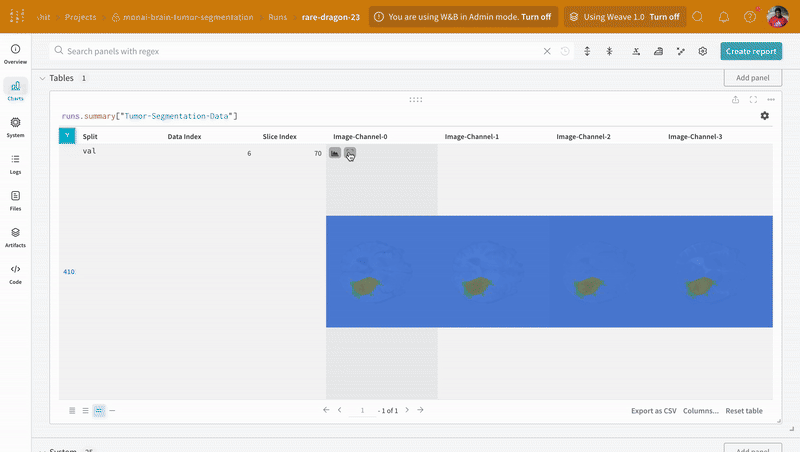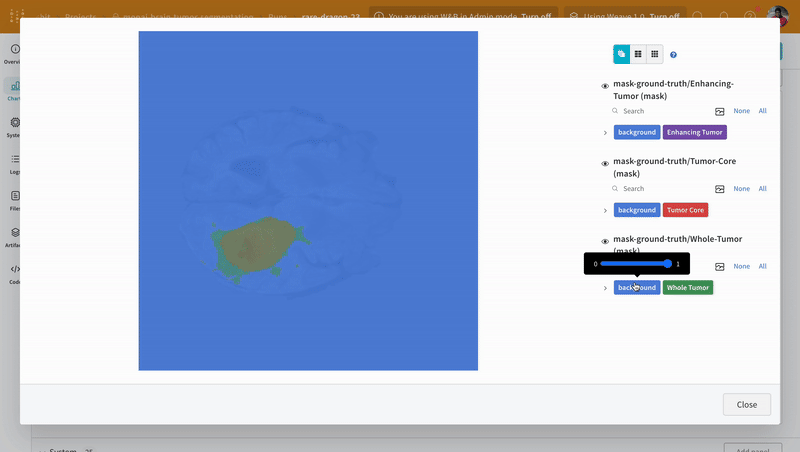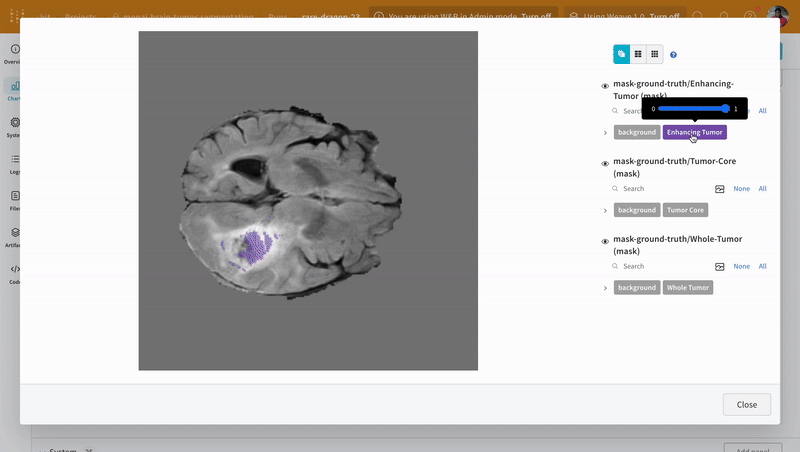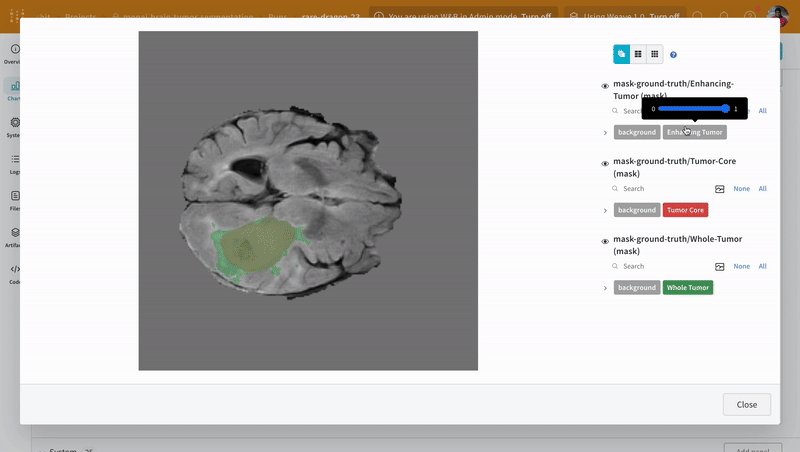
注意: データセットのラベルはクラス間で重ならないマスクで構成されています。オーバーレイはラベルをオーバーレイ内の個別のマスクとしてログします。
データのロード
データセットからデータをロードするための PyTorch DataLoaders を作成します。 DataLoaders を作成する前に、train_dataset の transform を train_transform に設定して、トレーニング用のデータを前処理および変換します。
モデル、損失、およびオプティマイザーの作成
このチュートリアルでは、 3D MRI brain tumor segmentation using auto-encoder regularization に基づいたSegResNet モデルを作成します。SegResNet モデルは、 monai.networks API の一部として PyTorch モジュールとして実装されています。これはオプティマイザーと学習率スケジューラともに利用可能です。
monai.losses API を使用してマルチラベル DiceLoss として定義し、それに対応するダイスメトリクスを monai.metrics API を使用して定義します。
トレーニングと検証
トレーニングの前に、トレーニングと検証の実験管理を追跡するためにwandb.log() でログを取るメトリクスプロパティを定義します。
標準的な PyTorch トレーニングループの実行
wandb.log で計装することで、トレーニングと検証プロセスに関連するメトリクスすべてを追跡するだけでなく、W&Bダッシュボード上のすべてのシステムメトリクス(この場合はCPUとGPU)も追跡できます。
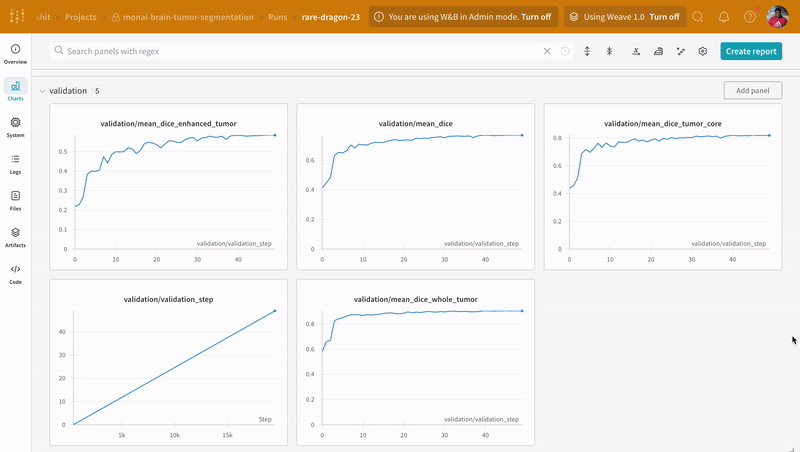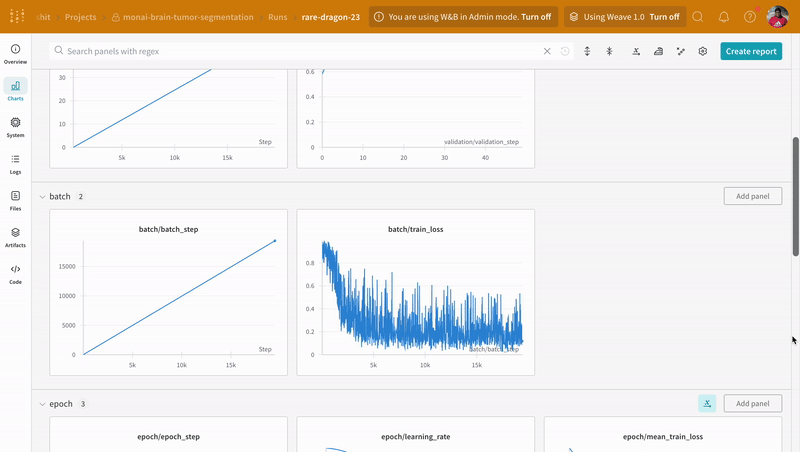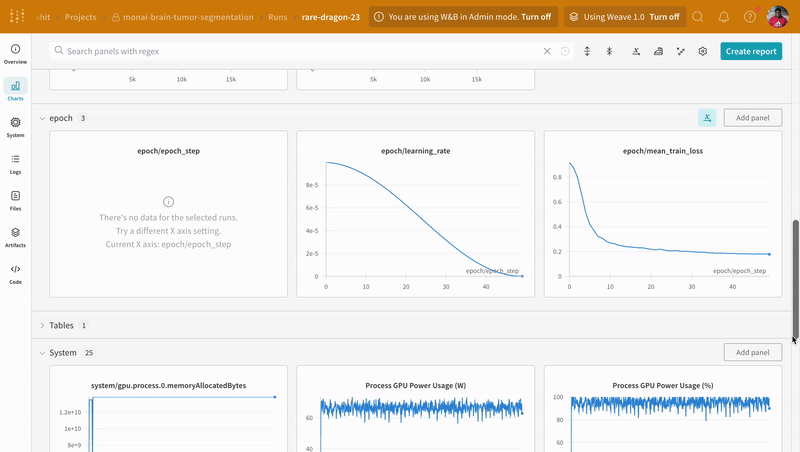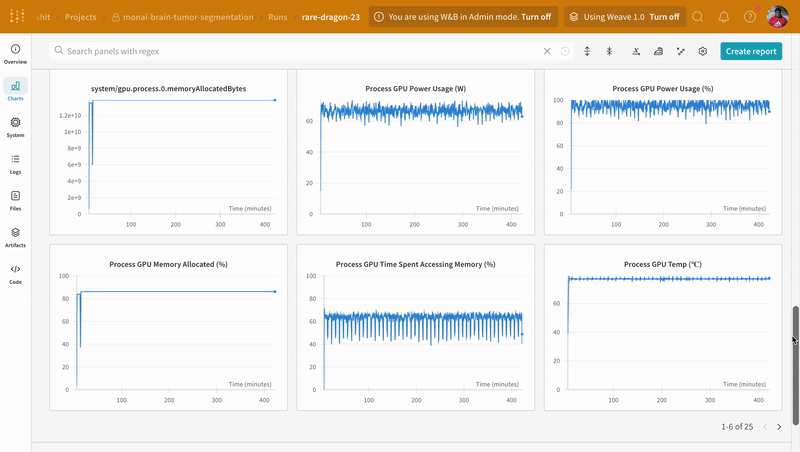
推論
アーティファクトインターフェースを使用して、このケースでは平均エポック単位のトレーニング損失が最良のモデルチェックポイントであるアーティファクトのバージョンを選択できます。アーティファクト全体のリネージを探索し、必要なバージョンを使用することもできます。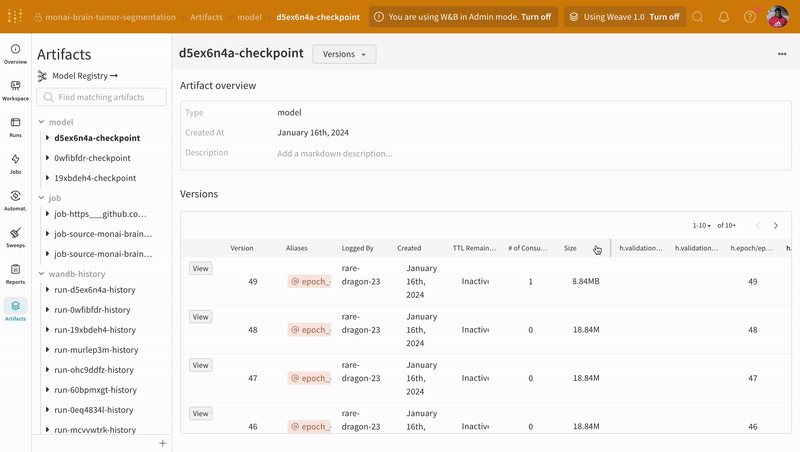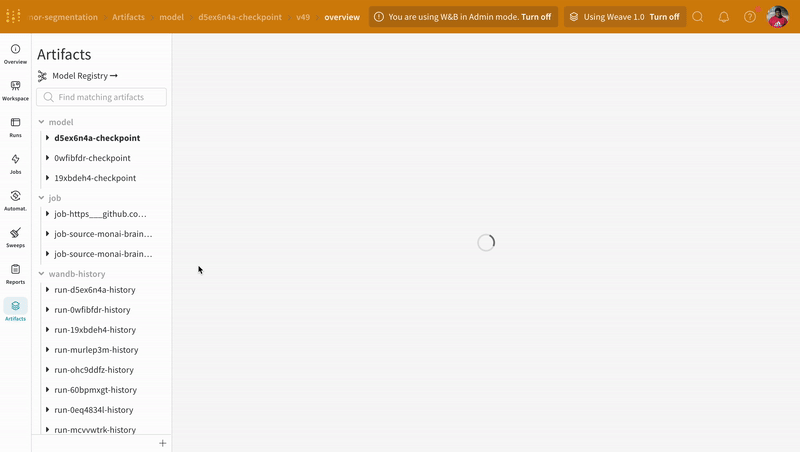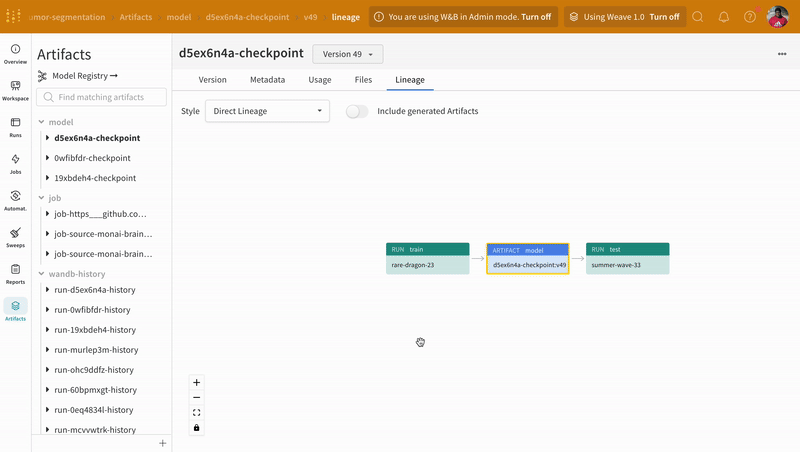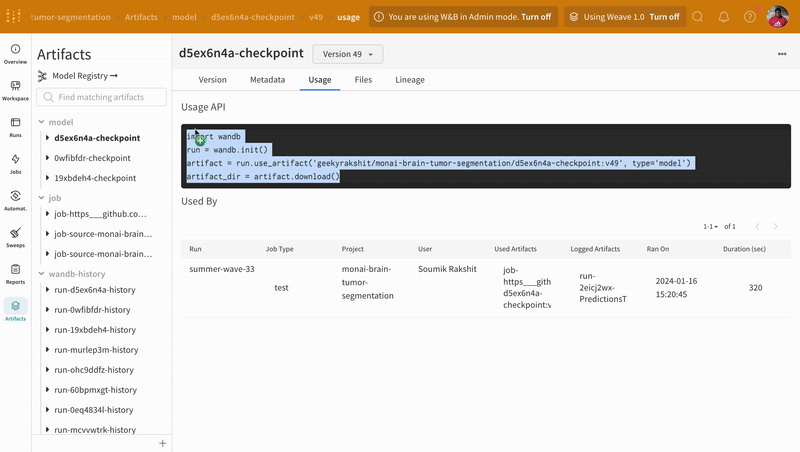
予測の可視化と正解ラベルとの比較
予測されたセグメンテーションマスクと対応する正解のセグメンテーションマスクをインタラクティブなセグメンテーションマスクオーバーレイを使用して視覚化するためのユーティリティ関数を作成します。