- W&B Run 을 초기화하고 재현성을 위해 run 과 관련된 모든 설정을 동기화합니다.
- MONAI transform API:
- 사전(dictionary) 형식 데이터에 대한 MONAI Transforms.
- MONAI
transformsAPI에 따라 새로운 transform을 정의하는 방법. - 데이터 증강(augmentation)을 위해 강도(intensity)를 무작위로 조정하는 방법.
- 데이터 로딩 및 시각화:
- 메타데이터와 함께
Nifti이미지를 로드하고, 이미지 리스트를 로드하여 쌓기(stack). - 트레이닝 및 검증 속도를 높이기 위한 IO 캐시 및 transforms 캐싱.
wandb.Table과 W&B의 대화형 세그멘테이션 오버레이를 사용하여 데이터 시각화.
- 메타데이터와 함께
- 3D
SegResNet모델 트레이닝- MONAI의
networks,losses,metricsAPI 사용. - PyTorch 트레이닝 루프를 사용하여 3D
SegResNet모델 트레이닝. - W&B를 사용하여 트레이닝 실험 추적.
- W&B의 아티팩트로서 모델 체크포인트를 로그하고 버전 관리.
- MONAI의
wandb.Table과 W&B의 대화형 세그멘테이션 오버레이를 사용하여 검증 데이터셋의 예측값 시각화 및 비교.
설정 및 설치
먼저, MONAI와 W&B의 최신 버전을 설치합니다.W&B Run 초기화
실험 추적을 시작하기 위해 새로운 W&B Run 을 시작합니다. 적절한 설정(config) 시스템을 사용하는 것은 재현 가능한 기계학습을 위한 권장 사항입니다. W&B를 사용하여 모든 실험의 하이퍼파라미터를 추적할 수 있습니다.데이터 로딩 및 변환
여기서는monai.transforms API를 사용하여 멀티 클래스 라벨을 원-핫(one-hot) 형식의 멀티 라벨 세그멘테이션 작업으로 변환하는 커스텀 transform을 생성합니다.
Dataset
이 실험에 사용된 데이터셋은 http://medicaldecathlon.com/ 에서 제공됩니다. 멀티 모달 멀티 사이트 MRI 데이터(FLAIR, T1w, T1gd, T2w)를 사용하여 신경교종(Gliomas), 괴사/활성 종양, 부종을 세그멘테이션합니다. 데이터셋은 750개의 4D 볼륨(트레이닝 484개 + 테스트 266개)으로 구성됩니다.DecathlonDataset을 사용하여 데이터셋을 자동으로 다운로드하고 압축을 풉니다. 이는 MONAI CacheDataset을 상속받으므로 메모리 크기에 따라 cache_num=N을 설정하여 트레이닝용으로 N개의 항목을 캐싱하거나, 기본 인수를 사용하여 검증용으로 모든 항목을 캐싱할 수 있습니다.
참고:
train_dataset에 train_transform을 적용하는 대신 트레이닝 및 검증 데이터셋 모두에 val_transform을 적용합니다. 이는 트레이닝 전에 데이터셋의 양쪽 분할에서 샘플을 시각화하기 위함입니다.데이터 시각화
W&B는 이미지, 비디오, 오디오 등을 지원합니다. 풍부한 미디어를 로그하여 결과를 탐색하고 run, 모델 및 데이터셋을 시각적으로 비교할 수 있습니다. 데이터 볼륨을 시각화하려면 세그멘테이션 마스크 오버레이 시스템을 사용하세요. tables에 세그멘테이션 마스크를 로그하려면 테이블의 각 행에 대해wandb.Image 오브젝트를 제공해야 합니다.
의사 코드(pseudocode) 예시는 다음과 같습니다:
wandb.Table 오브젝트 및 일부 관련 메타데이터를 가져와 W&B 대시보드에 로그될 테이블의 행을 채우는 간단한 유틸리티 함수를 작성합니다.
wandb.Table 오브젝트와 테이블을 구성하는 컬럼을 정의하여 데이터 시각화 결과로 채워질 수 있도록 합니다.
train_dataset과 val_dataset을 각각 루프 돌며 데이터 샘플에 대한 시각화를 생성하고, 대시보드에 로그할 테이블의 행을 채웁니다.
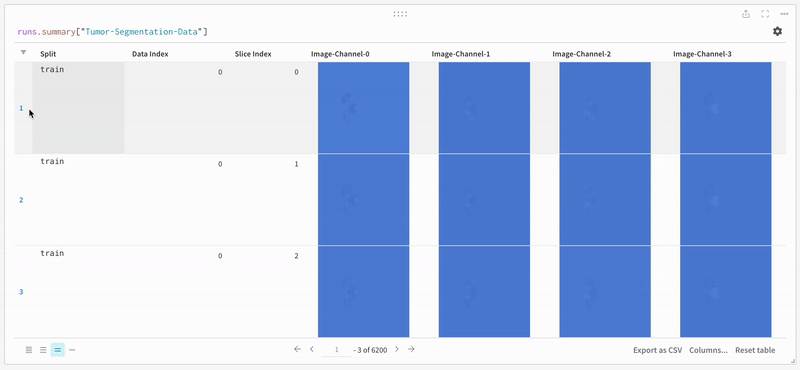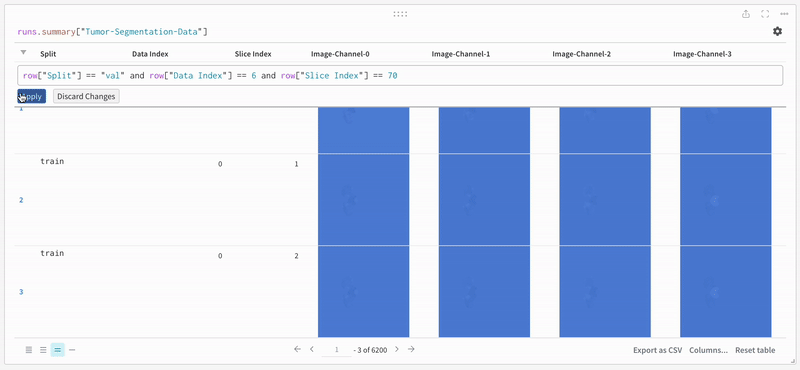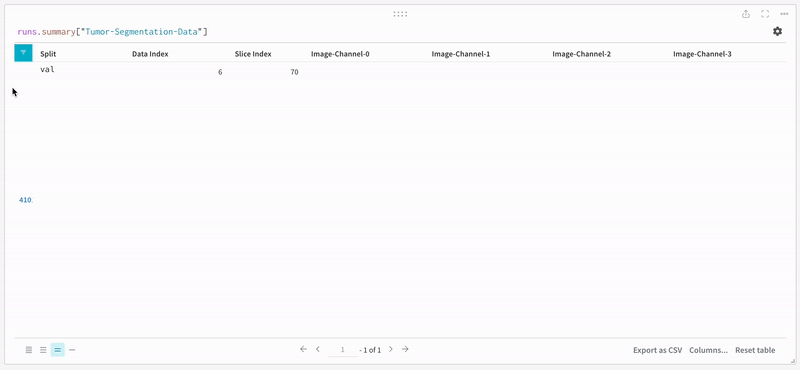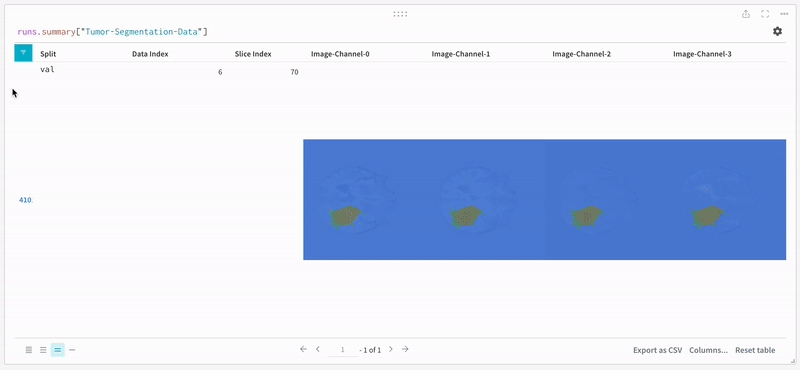
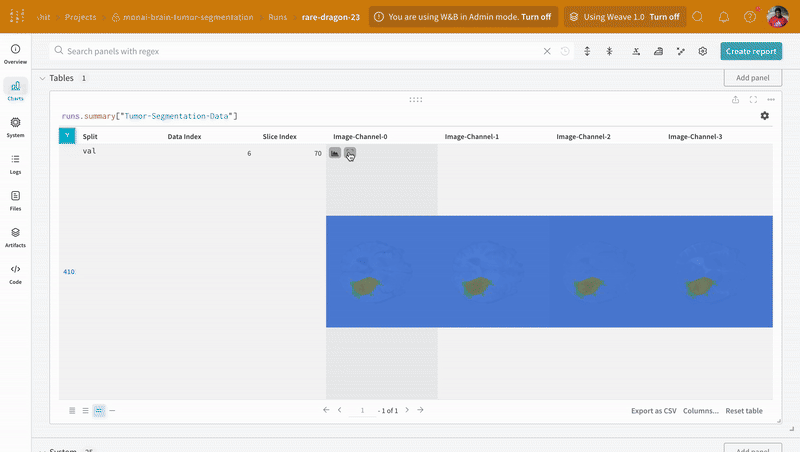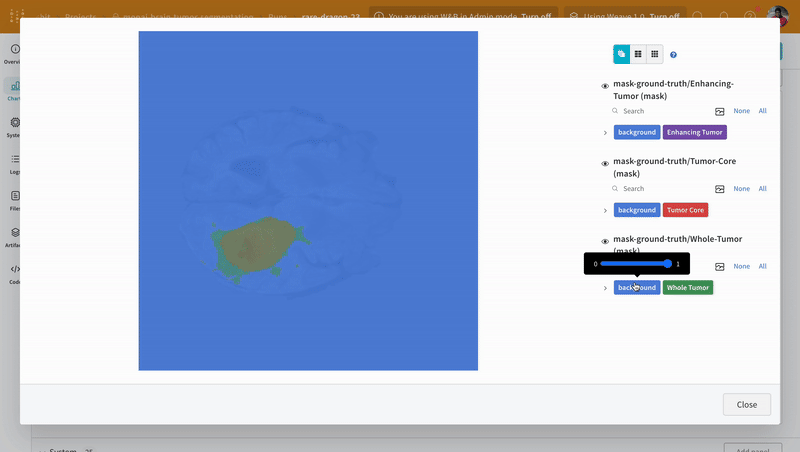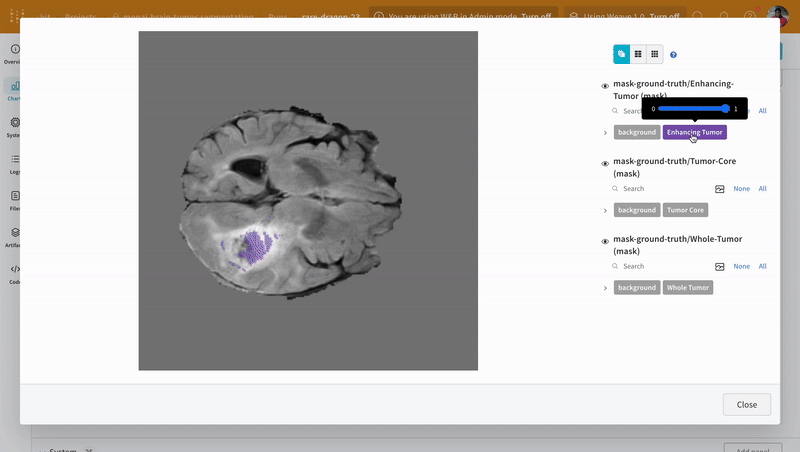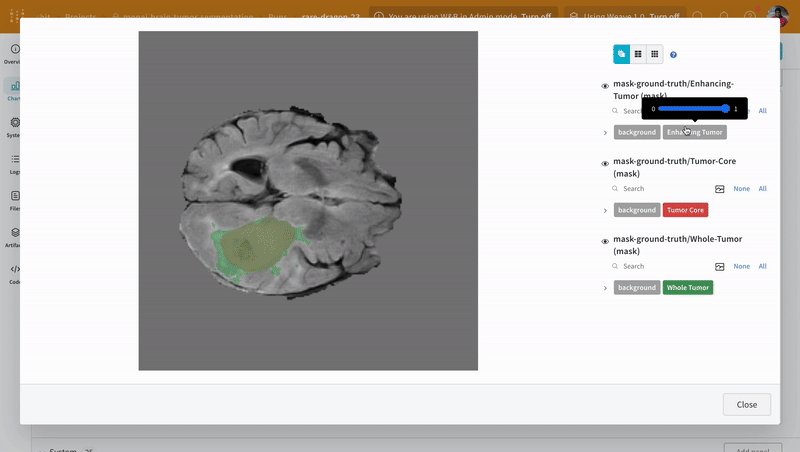
참고: 데이터셋의 라벨은 클래스 간에 겹치지 않는 마스크로 구성됩니다. 오버레이는 라벨을 오버레이 내의 개별 마스크로 로그합니다.
데이터 로딩
데이터셋에서 데이터를 로드하기 위한 PyTorch DataLoader를 생성합니다. DataLoader를 생성하기 전에train_dataset의 transform을 train_transform으로 설정하여 트레이닝을 위한 데이터를 전처리하고 변환합니다.
모델, 손실 함수 및 옵티마이저 생성
이 튜토리얼은 3D MRI brain tumor segmentation using auto-encoder regularization 논문을 기반으로SegResNet 모델을 생성합니다. SegResNet 모델은 옵티마이저 및 학습률 스케줄러와 함께 monai.networks API의 일부인 PyTorch 모듈로 구현되어 제공됩니다.
monai.losses API를 사용하여 손실 함수를 멀티 라벨 DiceLoss로 정의하고, monai.metrics API를 사용하여 해당 dice metrics를 정의합니다.
트레이닝 및 검증
트레이닝 전에 트레이닝 및 검증 실험 추적을 위해 나중에run.log()로 로그할 메트릭 속성을 정의합니다.
표준 PyTorch 트레이닝 루프 실행
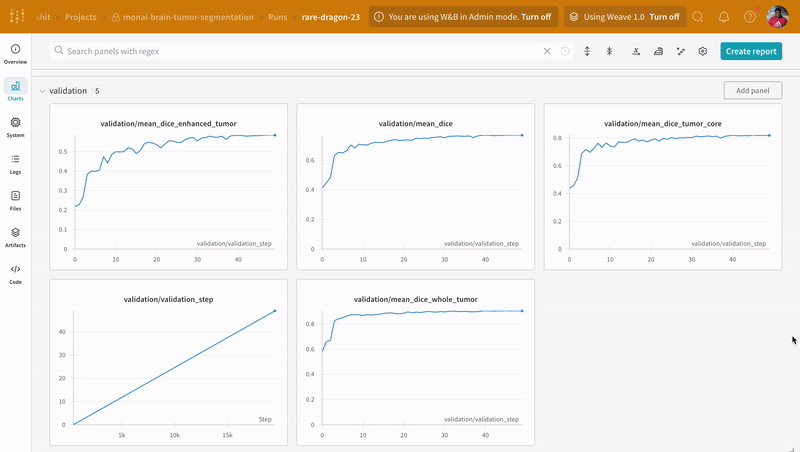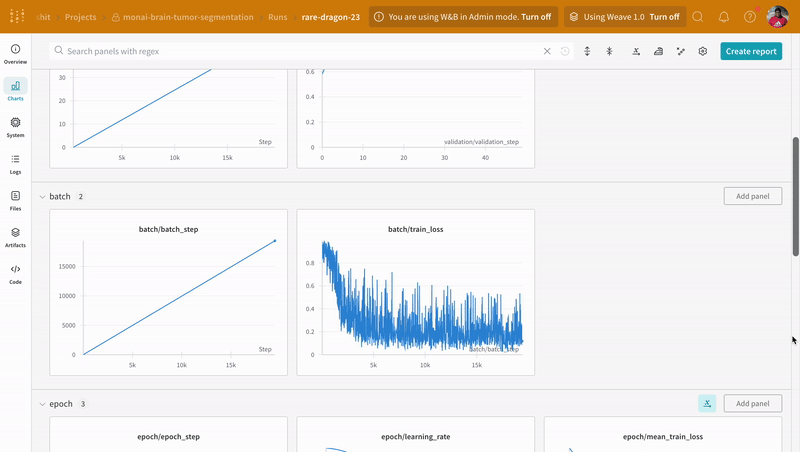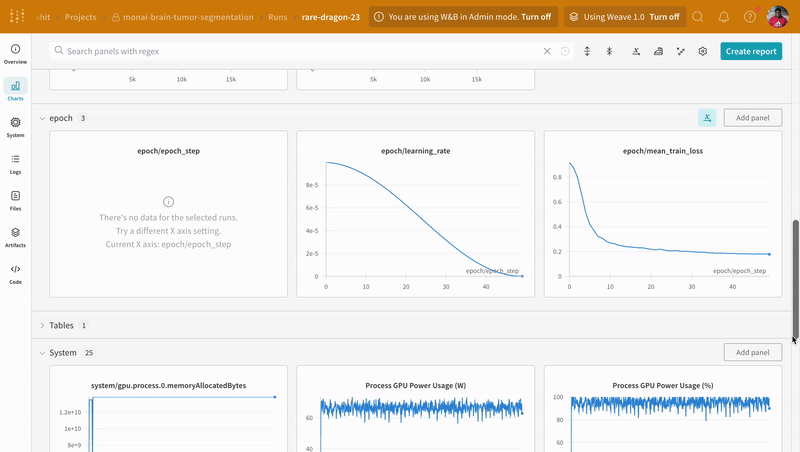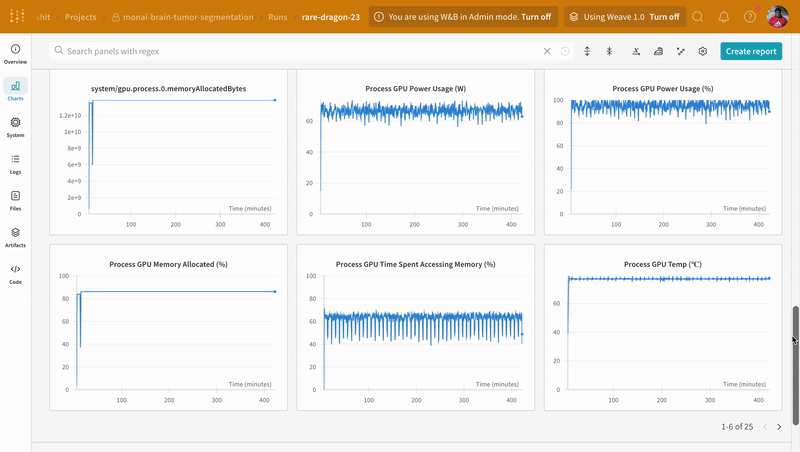
wandb.log로 계측하면 트레이닝 및 검증 프로세스와 관련된 모든 메트릭을 추적할 수 있을 뿐만 아니라, 모든 시스템 메트릭(이 경우 CPU 및 GPU)도 W&B 대시보드에 로그됩니다.
추론
아티팩트 인터페이스를 사용하여 어떤 버전의 아티팩트가 최상의 모델 체크포인트인지 선택할 수 있습니다(이 경우 에포크별 평균 트레이닝 손실 기준). 또한 아티팩트의 전체 계보(lineage)를 탐색하고 필요한 버전을 사용할 수 있습니다.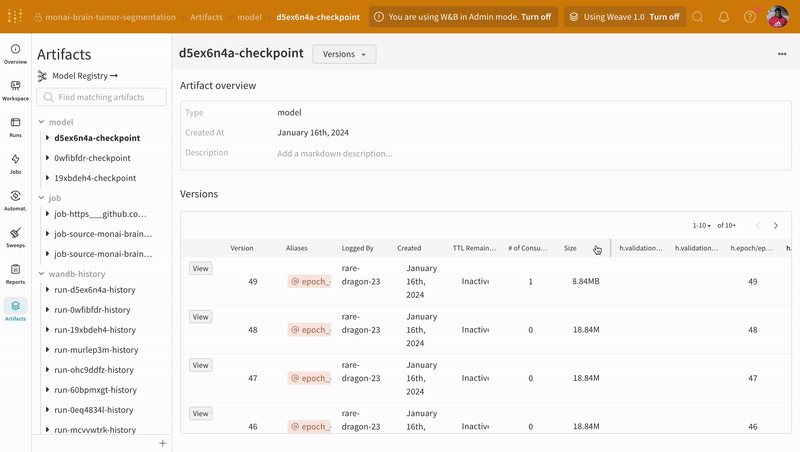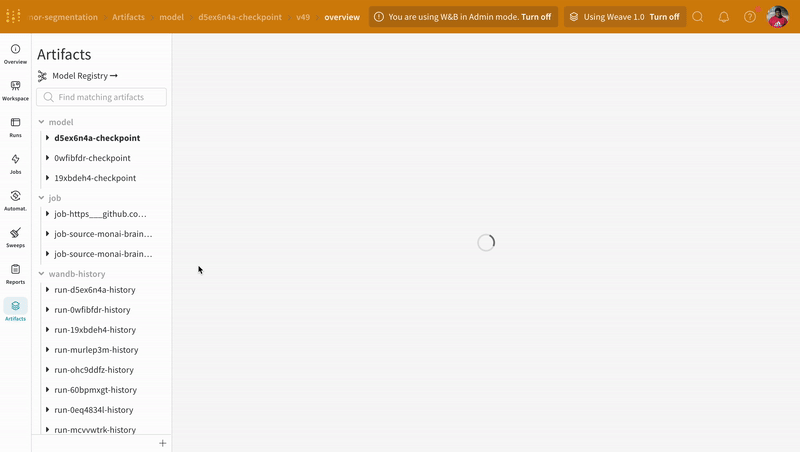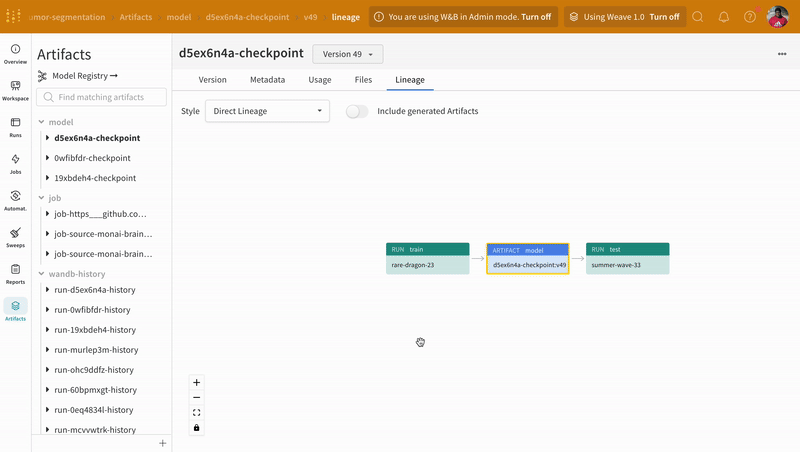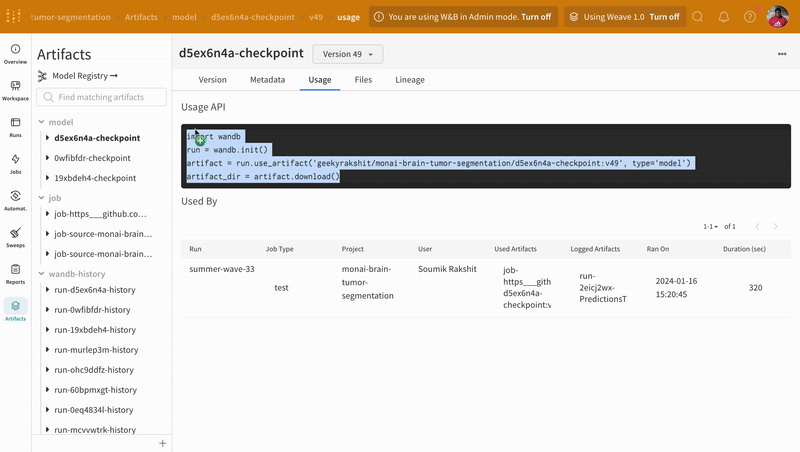
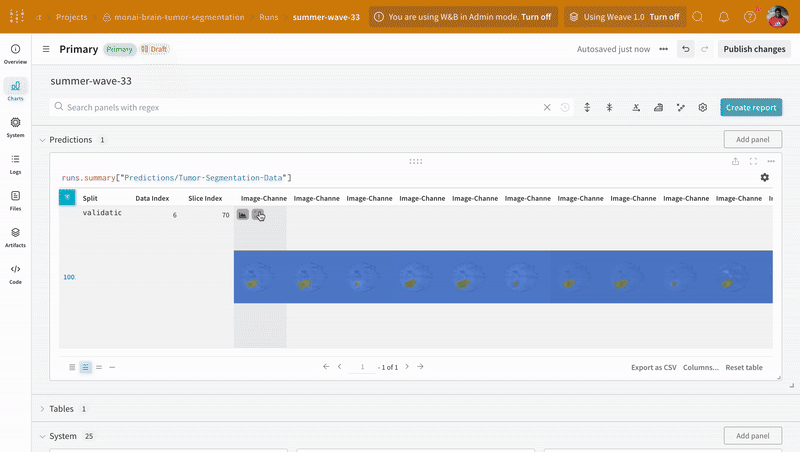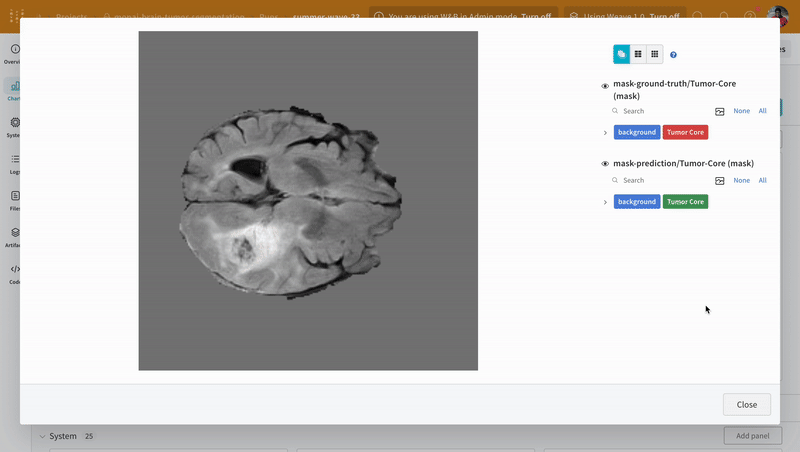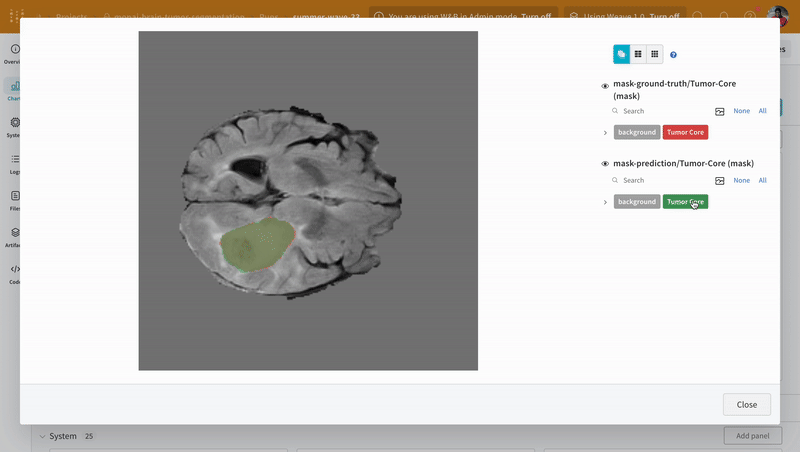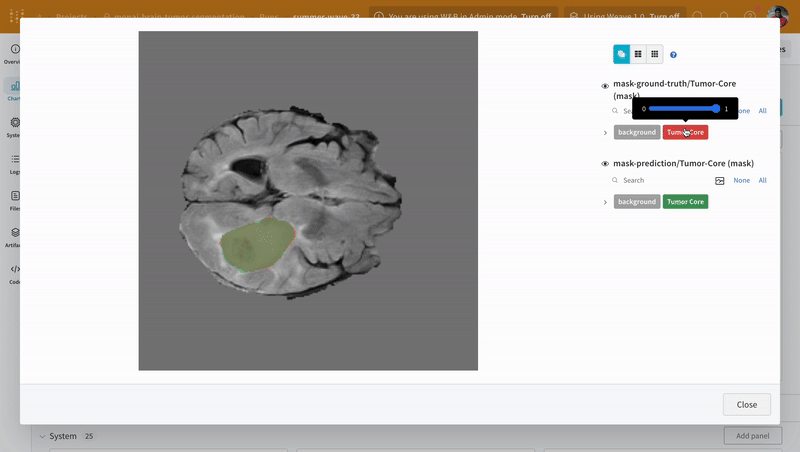
예측값 시각화 및 그라운드 트루스 라벨과 비교
대화형 세그멘테이션 마스크 오버레이를 사용하여 사전학습된 모델의 예측값을 시각화하고 해당 그라운드 트루스 세그멘테이션 마스크와 비교하는 또 다른 유틸리티 함수를 생성합니다.